본 포스팅에서는 model based novelty detection 방법인 Auto-encoder,1-SVM, SVDD, Isolation Forest 에 대해 자세히 다루겠습니다. 글의 전체적인 내용은 고려대학교 강필성 교수님의 Business-Analytics 강의를 참고하였음을 밝힙니다.
Auto Encoder
오토 인코더는 신경망 기반의 비지도 학습 모델로, 오토인코더를 거쳐나온 출력값이 입력값과 최대한 비슷해 지도록 하는 것을 목표로 학습합니다. 이때, 입력값의 차원보다 신경망 뉴런의 갯수가 크거나 같을 경우 학습의 의미가 없어집니다. 입력값을 그대로 받아서 내보내는것이 더 압축된 값이기 때문입니다. 학습의 결과, 따라서 학습의 결과는 더 적은수의 값들로 원래의 값을 복원할 수 있는 압축의 효과를 얻을 수 있습니다. 구조는 아래그림과 같으며, 신경망 두개를 거꾸로 이어붙인 형태입니다.
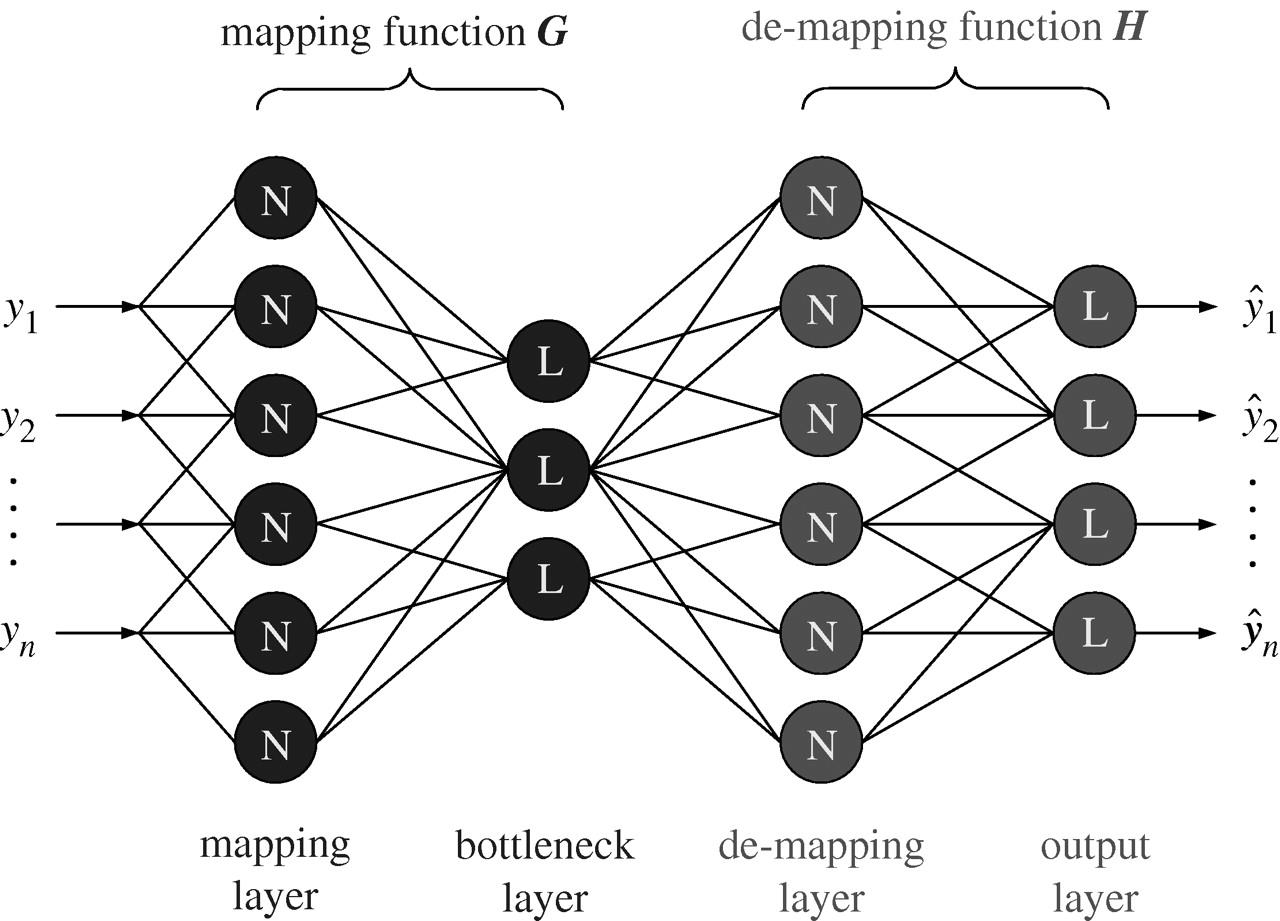
앞에 있는 뉴럴네트워크는 인코더, 뒤에 붙은 네트워크는 디코더가 됩니다.
인코더를 통해서 입력 데이타에 대한 특징을 추출해내고, 이 결과를 가지고 뉴럴 네트워크를 역으로 붙여서 원본 데이타를 생성해냅니다.

이 과정에서 입력과 출력값이 최대한 같아지도록 튜닝함으로써, Feature를 잘 추출할 수 있게 하는것이 오토 인코더의 원리 입니다.
Novelty detection에 이를 활용하는 방법은 Normal data로 학습되어있는 오토인코더에 학습이 되지 않은 데이터가 들어가는 경우(Anormal data)디코더에 의해 제대로 복원이 되지 않는 가정을 바탕으로 합니다. 실제로 다수의 정상데이터로 학습시킨 오토인코더의 신경망의 파라미터들은 정상데이터를 최대한 잘 복원하도록 튜닝이 되어있는 상태이기 때문에, 익숙하지 않은 비정상데이터가 input 이 되는 경우에는 복원이 잘 되지 않습니다. 다만, input 대비 output 이 얼마나 다를때 비정상으로 판단할 것인가에 대한 임계치 설정이 필요하며 이는 실제데이터를 통한 설정이나, 통계에 의해 결정합니다.
Density-based 나 clustering-based 등의 많은 모델이 모든 데이터의 이상치 점수(Anormality score)를 계산하여 이상치인지 아닌지를 판단하고자 했다면, 앞으로 살펴볼 1-SVM 과 SVDD 는 데이터 공간에서 경계면을 만들어 그것을 기준으로 정상데이터인지, 아닌지를 구분해주는 알고리즘 입니다.
1-SVM
1-svm(one class SVM)
- Original Problem

1-svm 은 feature space에서 정상 데이터를 원점에서 가장 멀게 밀어내는hyperplane을 찾는 알고리즘 입니다. 직관적이지 않을수 있기 때문에, 위의 그림을 통해 다시 설명하겠습니다. 위의 그림에서 초록색으로 칠해진 데이터만 정상임을 가정했을때, 굵게 그어진 원점에서 가장 멀리 떨어진hyperplane을 그어 그 hyper plane이 정상과 비정상을 구별하는 thershold가 되도록 하고 싶은것이 1-svm의 아이디어 입니다. 다만, 우리의 앞선 가정과는 달리 실제로는 위에 그려진 모든 데이터가 정상데이터이고, 그렇기 때문에 정상데이터 모두를 hyperplane 밖에 위치 시키려면 $\frac{b}{\lVert w\rVert}$가 작아질수 밖에 없습니다. 따라서 이런 데이터들에 대해서는 페널티($\xi$)를 주어, objective function 에 추가시켜, 마진을 최대화하도록 하되, 너무 많은 페널티는 주지 않도록하는 optimization 문제를 정의합니다. 아래에 있는 식이 위의 설명을 수식화한 식입니다.
(그림에서의 b는 식에서의 $\rho$와 같습니다.)
decision funciton
새로운 데이터를 hyperplane 의 식에 대입하여, 그 값이 +면 정상데이터, -면 비정상데이터로 분류하겠다는 의미입니다.
- primal Lagrangian problem
lagrangian multiplier를 이용하여, 원래의 fomulation을 다음과 같이 변형할 수 있습니다.
KKT condition
위의 식에서 우리가 구해야할 미지수가 무엇인지 생각해보겠습니다. $l,\mathbf{X_i}$는 데이터가 주어지면 알 수 있는 값들입니다. 따라서 우리가 식으로 부터 hyperplane 을 도출하고자 할 때, 구해야하는 미지수는 $\mathbf{W,\xi,\rho}$ 입니다. 따라서 목적식을 이 3개의 미지수에 대해서 편미분을 하여 다음과 같은 KKT condition 을 유도할 수 있습니다.
- Dual Lagrangian problem
(2),(3),(4)의 식을 (1)식에 대입하면,
이고, 이를 더 간단하게 정리하면,
로 바꿀수 있습니다. dual 은 간단히 설명하면, primal 의 목적식이 가질수 있는 값이 특정 조건하에서 항상 다른 목적식(dual)보다 크다는것을 보장 할 때, 이 dual을 최대화 하는 최적화 문제를 풀게 되면 primal 의 최소값을 구할 수 있다는 것이 아이디어 입니다. 여기서 한가지 중요한점은 primal의 최소값과 dual 의 최대값이 항상 같음을 보장하지는 않지만, 위에서 보았던 KKT condition을 만족하면 두 값은 같아지게 됩니다.

(6) 식에서 $\mathbf{\Phi(\mathbf{X_i})}\cdot\mathbf{\Phi(\mathbf{X_j})}$ 의 의미는 input space 에서 feature space로 $\Phi$가 각 $X_i,X_j$를 매핑하고 또 그것을 내적한다는 의미입니다. 이는 고차원으로 데이터를 매핑시켜 내적하는 상당한 연산을 요구하며 매핑함수 $\Phi$ 를 찾아야한다는 단점이 있습니다. 따라서 같은 값을 내는 Kernel function $\mathbf{K()}$을 정의하면 위와 같은 문제를 해결 할 수 있습니다. 즉 $\mathbf{\Phi(\mathbf{X_i})}\cdot\mathbf{\Phi(\mathbf{X_j})}=\mathbf{K(\mathbf{X_i,X_j})}$ 를 만족한다면 한번에 내적값을 구할수 있고, 따라서 (6)번 식은 다음과 같이 바꿔쓸 수 있습니다. 이를 kernel trick 이라고 합니다.
complementary slackness 에 의하여 $\alpha_i(\mathbf{W}\cdot\boldsymbol{\Phi}(\mathbf{X_i})-\rho+\xi_i)=0 \; , \beta_i\xi_i=0$ (*) 을 만족해야 합니다. 주의해야 할점은 각 식의 두항이 동시에 0이 될 수 없다는 것 입니다.
첫번째 경우 $\alpha_i=0$인 경우는 $\mathbf{W}\cdot\boldsymbol{\Phi}(\mathbf{X_i})-\rho+\xi_i\ne0$을 의미하기 때문에, 이 데이터 포인트는(i) support vector 가 아닙니다.
두번째 경우 $\alpha=\frac{1}{\nu l}$ 을 만족하는 경우, $\mathbf{W}\cdot\boldsymbol{\Phi}(\mathbf{X_i})-\rho+\xi_i=0$을 의미하기 때문에 support vector 이며, (3)식에 의해 $\beta_i=0$ 을 만족하게 되어 (*)에 의해 $\xi_i>0$ 을 만족합니다. 이는 이 데이터 포인트가 hyperplane 밖의 support vector 임을 의미합니다.
세번째 경우 $0<\alpha_i<\frac{1}{\nu l}$ 인경우, 마찬가지로 이 데이터포인트는 support vector 이고, (3)식에 의해, $\beta>0$ 을 만족하며, 이는 (*)에 의해 $\xi_i=0$ 즉 hyperplane 위의 support vector 임을 의미합니다.

-
hyperplane 밖에 있을수 있는 최대한의 support vectors의 갯수를 정해주는 비율 $\because \alpha_i\le\frac{1}{\nu l}$ 이므로 $\alpha_i$의 최대값은 $\frac{1}{\nu l}$입니다. 그런데 (4)에서 $\sum_{i=l}^l\alpha_i=1$을 만족해야 하므로 $\alpha=\frac{1}{\nu l}$를 만족하는 support vector들, 다시말해 위에서 설명한 hyperplane 밖에 있는 support vector 들의 최대갯수는 $\nu l$개 입니다.
-
데이터가 가져야하는 최소한의 support vector의 갯수를 정해주는 비율. $\because$ 위에 설명한 1번에 연장선에서 설명할 수가 있는데, $\alpha_i$들이 모두 $\frac{1}{\nu l}$인경우가 support vector의 전체 갯수는 가장 적은 경우라고 말할 수 있습니다. 하나라도 $\alpha_i<\frac{1}{\nu l}$인 i가 존재한다면, $\sum_{i=1}^l\alpha_i=1 $를 만족시키기위해 그 차이를 메꾸기 위한 nonzero 인 $\alpha_i$ 들이 존자해야하기 때문입니다.
def linear_kernel(X1, X2):
return X1.dot(X2.T)
def polynomial_kernel(X1, X2):
return (1 + X1.dot(X2.T)) ** 2
def gaussian_kernel(X1, X2):
return np.exp(-(X1 * X1)).dot(np.exp(-(X2 * X2)).T) * np.exp(2 * X1.dot(X2.T))
def one_svm(scope, X, y, args):
xmin, xmax, ymin, ymax = scope
V, kernel = args
l = len(X)
C = 1 / (l * V) # C 를 1/nu*l 로 정의
alpha = [0] * l
kX = kernel(X, X)
cons = ({'type': 'eq', 'fun': lambda alpha: np.sum(alpha) - 1}) # 제약식 정의
bnds = [[0, C]] * l
def target(alpha):
return 1 / 2 * (alpha).dot(kX).dot((alpha).T) #1-svm 에서의 목적식 정의(dual)
res = minimize(target, alpha, bounds=bnds, constraints=cons)
alpha_ = np.float16(res.x) # 너무 정교한 계산을 할 경우에 0을 0으로 저장하지 않는 경우가 발생할 수 있기 때문에 float16으로 변환
SVI, = np.where(alpha_ != 0) # 알파값이 0이 아닌 것들 (support vector)들의 index 추출
svs = X[SVI] #index 를 이용하여 X 값들을 추출.
MSVI = SVI[alpha_[SVI] < C] # support vector 중에 0<알파<C 인것들 -> on the hyperplane 의 index 찾아내기
msvs = X[MSVI]
plt.scatter(svs[:, 0], svs[:, 1], s=180, facecolors='#FFAAAA') # hyperplane 밖에 있는 정상 데이터 표시
plt.scatter(msvs[:, 0], msvs[:, 1], s=180, facecolors='#AAAAAA') # hyperplane 위에 있는 정상데이터 표시
plt.title("V = {}, {}".format(V, kernel.__name__), fontsize=16)
x0s = np.linspace(xmin, xmax, 200) # 등고선을 그리기 위한 x값 지정
x1s = np.linspace(ymin, ymax, 200) # 등고선을 그리기 위한 y값 지정
x0, x1 = np.meshgrid(x0s, x1s)
X_points = np.c_[x0.ravel(), x1.ravel()]
def prediction(X_points):
rhos = (alpha_).dot(kernel(X, msvs))
print(rhos)
rho = rhos[0]
result = (alpha_).dot(kernel(X, X_points)) - rho # decision function 정의
result[result >= 0] = 1
result[result < 0] = -1
return result
y_pred = prediction(X_points).reshape(x0.shape)
plt.contourf(x0, x1, y_pred, cmap=plt.cm.brg, alpha=0.2)
virginica = (raw_y == 2) # 데이터 지정.
y = raw_y[virginica]
for kernel in [linear_kernel, polynomial_kernel, gaussian_kernel]:
for V in [0.1, 0.05]:
plt.figure(figsize=(12,2.7))
try:
X = raw_X[virginica]
plot_boundary(one_svm, [4, 8, 0, 5], X, y, V, kernel)
except:
scaled_X = scaler.fit_transform(raw_X[virginica])
plot_boundary(one_svm, [-3, 3, -3, 3], scaled_X, y, V, kernel)
plt.show()
.JPG?raw=true)
.JPG?raw=true)
.JPG?raw=true)
Support Vector Data Description(SVDD)
SVDD 는 1-SVM 과는 달리 normal data 를 둘러싸는 가장작은 초구를 만들어 새로운 데이터가 초구를 기준으로 어디에 위치하는지를 통해 이상치인지 아닌지에 대한 판단을 하고자 하는 알고리즘 입니다. 다음 그림은 1-SVM 과 SVDD 를 직관적으로 각각의 아이디어를 나타낸 그림입니다.

SVDD 에서는 목적식을 통해 데이터를 둘러싸는 가장 작은 초구의 반지름과, 중심을 찾고자 합니다. primal problem 을 정의하고 제약식을 통해 primal Lagrangian problem 을 만들고 이를 통해 dual problem 을 만들어 최적화를 문제를 푸는 일련의 과정은 1-SVM 과 매우 유사하기 때문에 자세한 설명은 생략하도록 하겠습니다.
primal problem
Decision funcion
primal lagrangian problem
KKT condition
dual lagrangian problem
(8),(9),(10)의 식을 (7)번 식에 대입하면 dual problem 을 얻을 수 있습니다.
마지막으로 정리하면 다음과 같습니다.
이를 Minimization 문제로 바꾸기 위해서는 단순히 -1을 곱하면 됩니다. 최종적으로 dual problem 은
입니다.
위에서 설명한 kernel trick 을 이용하연

두번째, $\alpha=C$ 인경우,hypersphere 밖의 support vector 입니다. $\because \lVert \mathbf{\Phi(\mathbf{X_i})-\mathbf{a}} \rVert^2 - R^2+\xi_i=0$을 만족하고,$C-\alpha_i-\beta_i=0\quad(10)$에 의해, $\beta_i=0$을 만족합니다. 따라서 $\xi_i>0$ 이고 이는 페널티를 먹는 hypersphere 밖의 support vector 입니다.
세번째, $0<\alpha_i <$C 는 hypersphere 위의 support vetor 입니다. $\because$ 두번째 경우에서 $\beta_i\ne0$ 이고 $\xi_i=0$ 이기 때문입니다.
1-SVM 과 SVDD 가 같은이유
$\lVert w \rVert^2=1 $ 이고 모든 데이터를 unit norm vector 로 바꾸는경우에 두가지 방법은 완전히 같은 알고리즘 입니다.
이경우 1-SVM의 objective function 은:
으로 바뀝니다.(앞에 남는 $\frac{1}{2}$는 상수이기 때문에 무시해도 됩니다.)
$\lVert w \rVert$ 에 관한 제약조건이 추가 되었기 때문에 목적함수의 미지수는 현재 $\rho , \xi$ 입니다.
또 앞에서 말했던것처럼 모든 데이터를 unit norm vector 로 바꿀때, 정보의 손실을 없애기 위해 다음과 같은 방법을 제시할 수 있다.
다시 svdd의 목적함수로 되돌아가 보면,
위의 식에서 모든 벡터가 unit norm vector 로 변환되었다고 하자. 따라서 svdd의 목적함수와 제약식을 다음과 같이 쓸 수 있습니다.
또한, 제약식은 다음과 같이 풀어 쓸 수 있습니다.
따라서 svdd 의 목적함수와 제약식을 다음과 같이 다시 쓸 수 있습니다.
여기서, $\mathbf{W}=\mathbf{a’}, \rho=\frac{1}{2}(2-R’^2), \frac{1}{\nu l}=C’, \xi_i=\frac{1}{2}\xi’_i,\; \forall i $ 로 정의하면, 다음과 같은 optimization problem 이 얻어집니다.
이는 (1) 에서 우리가 보였던 식과 같습니다. 따라서 $\lVert w \rVert=1$인 경우, svdd 와 1-svm은 같은 알고리즘 입니다.
def svdd(scope, X, y, args):
xmin, xmax, ymin, ymax = scope
V, kernel = args
l = len(X)
C = 1 / (l * V)
alpha = [0] * l
kX = kernel(X, X)
cons = ({'type': 'eq', 'fun': lambda alpha: sum(alpha) - 1}) # constarint 정의
bnds = [[0, C]] * l # 바운드 정의
def target(alpha):
return alpha.dot(kX).dot(alpha.T) - alpha.dot(np.diag(kX)) # SVDD 의 목적식 입력.
res = minimize(target, alpha, bounds=bnds, constraints=cons)
alpha_ = np.float16(res.x)
SVI = np.where(alpha_ != 0)[0] # 알파 값이 0이 아닌것이 support vector이기 때문. 따라서 그것들의 index 저장
svs = X[SVI] # 위에서 저장한 index 값을 이용하여 그 index의 X 값 추출.
MSVI = SVI[alpha_[SVI] < C] # on the hypersphere 의 index 추출
msvs = X[MSVI] # 위에서 추출한 index 값들을 이용하여 X 값 추출.
plt.scatter(svs[:, 0], svs[:, 1], s=180, facecolors='#FFAAAA') # hypersphere 밖에 있는 support vector 들을 색칠
plt.scatter(msvs[:, 0], msvs[:, 1], s=180, facecolors='#AAAAAA') # hypersphere 에 있는 support vector 들을 색칠
plt.title("V = {}, {}".format(V, kernel.__name__), fontsize=16)
x0s = np.linspace(xmin, xmax, 50) # 등고선을 그리기 위한 x축값 지정
x1s = np.linspace(ymin, ymax, 50) # 등고선을 그리기 위한 y 축값 지정
x0, x1 = np.meshgrid(x0s, x1s)
X_points = np.c_[x0.ravel(), x1.ravel()]
def prediction(X_points):
rss = (np.diag(kernel(msvs, msvs)) +
alpha_.dot(kX).dot(alpha_.T) - 2 * alpha_.dot(kernel(X, msvs)))
print(rss)
rs = rss[0]
result = rs - (np.diag(kernel(X_points, X_points)) +
alpha_.dot(kX).dot(alpha_.T) -
2 * alpha_.dot(kernel(X, X_points)))
result[result >= 0] = 1
result[result < 0] = -1
return result
y_pred = prediction(X_points).reshape(x0.shape)
plt.contourf(x0, x1, y_pred, cmap=plt.cm.brg, alpha=0.2)
virginica = (raw_y == 2)
scaled_X = scaler.fit_transform(raw_X[virginica])
y = raw_y[virginica]
for kernel in [linear_kernel, polynomial_kernel, gaussian_kernel]:
plt.figure(figsize=(12,2.7))
plot_boundary(svdd, [-3, 3, -3, 3], scaled_X, y, 0.05, kernel)
plt.show()
Isolation Forest
Isolation forest 알고리즘은 위에서 소개한 svdd,1-svm 과는 달리 데이터별 novelty score를 계산하는 알고리즘 입니다.
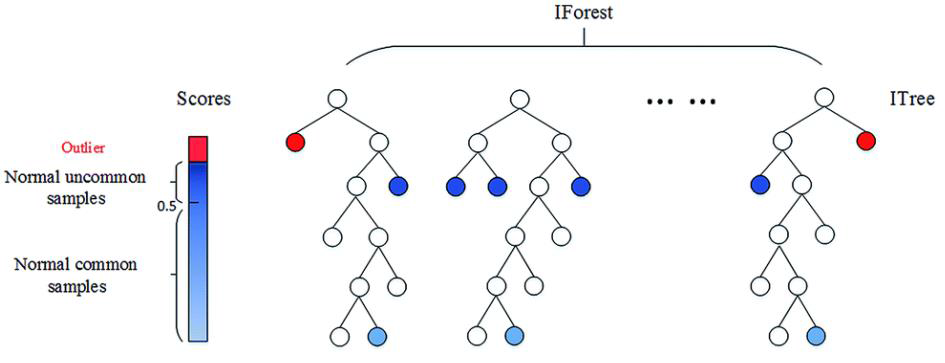
알고리즘의 아이디어는 랜덤하게 차원을 선택해서 임의의 기준으로 공간을 분할할때. 군집 내부에 있는 정상치 $x_i$의 경우 공간 내에 한 점만 남기고 완전히 고립시키려면 많은 횟수의 공간 분할을 수행해야 하지만, 군집에서 멀리 떨어진 이상치 $x_o$는 적은 횟수의 공간 분할만으로 고립시킬 수 있다는 것입니다.
공간분할은 차원과 기준 값으로 표현할 수 있으므로, 여러 번의 공간분할은 의사결정나무 (Decision Tree) 형태로 표현할 수 있습니다. 정상치일수록 완전히 고립시킬 수 있을 때까지 의사결정나무를 깊숙하게 타고 내려가야 합니다. 반대로 이상치의 경우, 의사결정나무의 상단부만 타더라도 고립될 가능성이 높습니다. 이런 특성을 이용하면 의사결정나무를 몇 회 타고 내려가야 고립되는가를 기준으로 정상치와 이상치를 분리할 수 있습니다.

Definition of Isolation forest
전체 dataset 에서 sampling 을 한 X dataset(n instance from d variate distribution) 을 추출하고, X 를 지속적으로 random하게 선택된 variable과 value 로 분할 하는 것입니다. 보통의 경우 sample data 의 갯수는 256개를 많이 이용합니다. 분할을 멈추는 경우는 다음과 같이 3가지 경우입니다.
-
인 경우
-
각 Terminal node에 있는 instance의 값들이 모두 같은 경우
-
Tree의 height가 정해진 값(hyper-parameter)가 된경우
Definition of Path length
-
Path length h(x)는 x 가 root node 에서 x가 속하는terminal node 까지 가면서 거치는 edge의 갯수입니다.
-
c(n)은 n개의 instance 를 이용하여 isolation tree를 구성할때, 정상데이터의 path length의 기댓값 입니다.
(c(n)=2H(n–1)-(2(n-1)/n),(H(i)=ln(i)+0.5772156649)
Definition of Novelty score
즉, 특정 데이터 포인트의 평균 path length(E[h(x)])가 매우 큰 경우 다시말해 isolation forest 의 가정에 의해 정상데이터일 확률이 높은 경우 s(x,n)의 값은 0에 가까워지며, 반대로 path length 가 매우 짧은 경우 다시말해 비정상 데이터일 확률이 높은경우 s(x,n) 의값은 1에 가까워집니다. 따라서 이를 score 처럼 활용 하여 새로운 데이터에 대한 s(x,n) 값으로 정상과 비정상 데이터를 구분하는 근거를 얻을 수 있습니다.

조금은 이해하기 난해할수도 있기 때문에 예시를 들어보겠습니다.
| column | A | B |
|---|---|---|
| Tree1 | 1 | 15 |
| Tree2 | 2 | 16 |
| Tree3 | 3 | 10 |
| … | … | … |
| Tree n | E[h(A)] | E[h(B)] |
위의 표는 데이터 A,B 의 각 tree 별 path length 를 구한 결과를 보여줍니다.
-
256개의 sample data로 구성되어 있는 Tree k(k=1,2,…n)에 대해서 A,B 의 path length를 구합니다.
-
그것을 평균을 내어 E[h(A)], E[h(B)]를 구합니다.
-
이를 이용하여 A,B 각각의 novelty score를 구합니다.
import pandas as pd
import numpy as np
import math
import random
%matplotlib inline
import random
from matplotlib import pyplot
import os
print(os.listdir("C:\\Users\korea\Desktop\creditcardfraud"))
class ExNode: #external(leaf) node정의
def __init__(self,size):
self.size=size
class InNode: #internal node 정의
def __init__(self,left,right,splitAtt,splitVal):
self.left=left
self.right=right
self.splitAtt=splitAtt
self.splitVal=splitVal
#isolation forest 정의
def iForest(X,noOfTrees,sampleSize):
forest=[]
hlim=math.ceil(math.log(sampleSize,2))
for i in range(noOfTrees):
X_train=X.sample(sampleSize)
forest.append(iTree(X_train,0,hlim))
return forest
def iTree(X,currHeight,hlim):
if currHeight>=hlim or len(X)<=1:
return ExNode(len(X))
else:
Q=X.columns
q=random.choice(Q)
p=random.choice(X[q].unique())
X_l=X[X[q]<p]
X_r=X[X[q]>=p]
return InNode(iTree(X_l,currHeight+1,hlim),iTree(X_r,currHeight+1,hlim),q,p)
def pathLength(x,Tree,currHeight): # pathlength 계산.
if isinstance(Tree,ExNode): # 만약 leaf node 면, 현재 length를 반환
return currHeight
a=Tree.splitAtt
if x[a]<Tree.splitVal: #내가 지정한 값보다 작은경우
return pathLength(x,Tree.left,currHeight+1)
else:
return pathLength(x,Tree.right,currHeight+1)
df=pd.read_csv("C:\\Users\korea\Desktop\creditcardfraud\creditcard.csv")
y_true=df['Class']
df_data=df.drop('Class',1)
sampleSize=10000 # score 를 매길때 sample 하는 data의 갯수/
ifor=iForest(df_data.sample(100000),10,sampleSize)
posLenLst=[]
negLenLst=[]
for sim in range(1000):
ind=random.choice(df_data[y_true==1].index)
for tree in ifor:
posLenLst.append(pathLength(df_data.iloc[ind],tree,0))
ind=random.choice(df_data[y_true==0].index)
for tree in ifor:
negLenLst.append(pathLength(df_data.iloc[ind],tree,0))
# path length 를 normal 과 abnormal 이 얼마나 차이가 나는지 비교.
bins = np.linspace(0,math.ceil(math.log(sampleSize,2)), math.ceil(math.log(sampleSize,2)))
pyplot.figure(figsize=(12,8))
pyplot.hist(posLenLst, bins, alpha=0.5, label='Anomaly')
pyplot.hist(negLenLst, bins, alpha=0.5, label='Normal')
pyplot.xlabel('Path Length')
pyplot.ylabel('Frequency')
pyplot.legend(loc='upper left')
 normal data가 압도적으로 긴 length 를 가지는것을 알수 있습니다.
normal data가 압도적으로 긴 length 를 가지는것을 알수 있습니다.


.JPG?raw=true)